Hinweis
Zum Ende springen, um den vollständigen Beispielcode herunterzuladen.
Bild-Neuberechnung#
Bilder werden durch diskrete Pixel dargestellt, denen Farbwerte zugewiesen sind, entweder auf dem Bildschirm oder in einer Bilddatei. Wenn ein Benutzer imshow mit einem Datenarray aufruft, ist es selten, dass die Größe des Datenarrays genau mit der Anzahl der dem Bild in der Abbildung zugewiesenen Pixel übereinstimmt, sodass Matplotlib die Daten oder das Bild neu berechnet oder skaliert, um sie anzupassen. Wenn das Datenarray größer ist als die Anzahl der im gerenderten Bild zugewiesenen Pixel, wird das Bild "down-sampled" (unterabgetastet) und Bildinformationen gehen verloren. Umgekehrt, wenn das Datenarray kleiner ist als die Anzahl der Ausgabepixel, erhält jeder Datenpunkt mehrere Pixel, und das Bild wird "up-sampled" (überabgetastet).
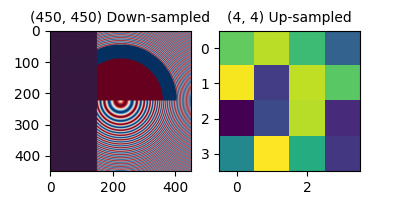
Im folgenden Diagramm hat das erste Datenarray die Größe (450, 450), wird aber durch weitaus weniger Pixel im Diagramm dargestellt und daher down-sampled. Das zweite Datenarray hat die Größe (4, 4) und wird durch weitaus mehr Pixel dargestellt und daher up-sampled.
import matplotlib.pyplot as plt
import numpy as np
fig, axs = plt.subplots(1, 2, figsize=(4, 2))
# First we generate a 450x450 pixel image with varying frequency content:
N = 450
x = np.arange(N) / N - 0.5
y = np.arange(N) / N - 0.5
aa = np.ones((N, N))
aa[::2, :] = -1
X, Y = np.meshgrid(x, y)
R = np.sqrt(X**2 + Y**2)
f0 = 5
k = 100
a = np.sin(np.pi * 2 * (f0 * R + k * R**2 / 2))
# make the left hand side of this
a[:int(N / 2), :][R[:int(N / 2), :] < 0.4] = -1
a[:int(N / 2), :][R[:int(N / 2), :] < 0.3] = 1
aa[:, int(N / 3):] = a[:, int(N / 3):]
alarge = aa
axs[0].imshow(alarge, cmap='RdBu_r')
axs[0].set_title('(450, 450) Down-sampled', fontsize='medium')
np.random.seed(19680801+9)
asmall = np.random.rand(4, 4)
axs[1].imshow(asmall, cmap='viridis')
axs[1].set_title('(4, 4) Up-sampled', fontsize='medium')
Die Methode imshow von Matplotlib verfügt über zwei Schlüsselwortargumente, mit denen der Benutzer steuern kann, wie die Neuberechnung durchgeführt wird. Das Schlüsselwortargument interpolation ermöglicht die Wahl des Kernels, der für die Neuberechnung verwendet wird, und ermöglicht entweder eine Anti-Aliasing-Filterung beim Down-Sampling oder eine Glättung von Pixeln beim Up-Sampling. Das Schlüsselwortargument interpolation_stage bestimmt, ob dieser Glättungs-Kernel auf die zugrunde liegenden Daten angewendet wird oder ob der Kernel auf die RGBA-Pixel angewendet wird.
interpolation_stage='rgba': Daten -> Normalisieren -> RGBA -> Interpolieren/Neuberechnen
interpolation_stage='data': Daten -> Interpolieren/Neuberechnen -> Normalisieren -> RGBA
Für beide Schlüsselwortargumente hat Matplotlib einen Standardwert "antialiased", der für die meisten Situationen empfohlen wird und unten beschrieben wird. Beachten Sie, dass sich dieses Standardverhalten je nachdem, ob das Bild down- oder up-sampled wird, unterschiedlich verhält, wie unten beschrieben.
Down-Sampling und mäßiges Up-Sampling#
Beim Down-Sampling von Daten möchten wir normalerweise Aliasing durch Glättung des Bildes entfernen und es dann unterabteilen. In Matplotlib können wir diese Glättung durchführen, bevor wir die Daten in Farben umwandeln, oder wir können die Glättung auf den RGB(A)-Bildpixeln durchführen. Die Unterschiede zwischen diesen werden unten gezeigt und mit dem Schlüsselwortargument interpolation_stage gesteuert.
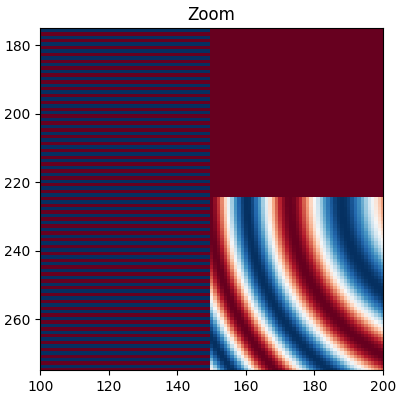
Die folgenden Bilder werden von 450 Datenpixeln auf ungefähr 125 Pixel oder 250 Pixel (je nach Anzeige) down-sampled. Das zugrunde liegende Bild hat abwechselnde +1, -1 Streifen auf der linken Seite und ein Muster mit variierender Wellenlänge (Chirp) im Rest des Bildes. Wenn wir hineinzoomen, können wir dieses Detail ohne Down-Sampling sehen
fig, ax = plt.subplots(figsize=(4, 4), layout='compressed')
ax.imshow(alarge, interpolation='nearest', cmap='RdBu_r')
ax.set_xlim(100, 200)
ax.set_ylim(275, 175)
ax.set_title('Zoom')
Wenn wir down-sampled, ist der einfachste Algorithmus, die Daten mittels Nearest-Neighbor-Interpolation zu dezimieren. Wir können dies entweder im Datenraum oder im RGBA-Raum tun
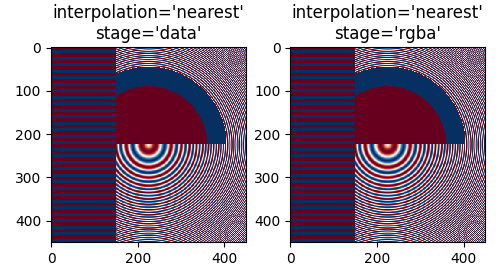
Die Nearest-Interpolation ist im Daten- und RGBA-Raum identisch, und beide weisen Moiré-Muster auf, da die hochfrequenten Daten down-sampled werden und als niederfrequente Muster erscheinen. Wir können die Moiré-Muster reduzieren, indem wir einen Anti-Aliasing-Filter auf das Bild anwenden, bevor es gerendert wird
fig, axs = plt.subplots(1, 2, figsize=(5, 2.7), layout='compressed')
for ax, interp, space in zip(axs.flat, ['hanning', 'hanning'],
['data', 'rgba']):
ax.imshow(alarge, interpolation=interp, interpolation_stage=space,
cmap='RdBu_r')
ax.set_title(f"interpolation='{interp}'\nstage='{space}'")
plt.show()
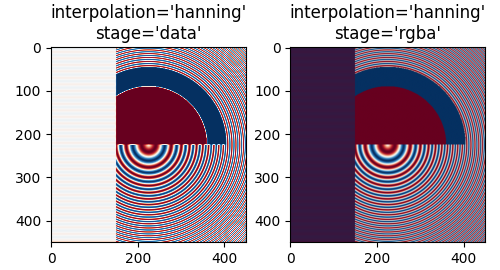
Der Hanning-Filter glättet die zugrunde liegenden Daten, sodass jedes neue Pixel ein gewichteter Durchschnitt der ursprünglichen zugrunde liegenden Pixel ist. Dies reduziert die Moiré-Muster erheblich. Wenn jedoch interpolation_stage auf 'data' gesetzt ist, führt dies auch zu weißen Bereichen im Bild, die nicht in den Originaldaten vorhanden sind, sowohl in den abwechselnden Bändern auf der linken Seite des Bildes als auch an der Grenze zwischen Rot und Blau der großen Kreise in der Mitte des Bildes. Die Interpolation auf der 'rgba'-Stufe hat einen anderen Artefakt, wobei die abwechselnden Bänder eine lilafarbene Nuance aufweisen; obwohl Lila nicht in der ursprünglichen Colormap vorhanden ist, ist es das, was wir wahrnehmen, wenn ein blaues und ein rotes Band nahe beieinander liegen.
Der Standardwert für das Schlüsselwortargument interpolation ist 'auto', das einen Hanning-Filter wählt, wenn das Bild um weniger als den Faktor drei down- oder up-sampled wird. Das Standard-Schlüsselwortargument interpolation_stage ist ebenfalls 'auto', und für Bilder, die um weniger als den Faktor drei down- oder up-sampled werden, wird standardmäßig 'rgba'-Interpolation verwendet.
Anti-Aliasing-Filterung ist auch beim Up-Sampling erforderlich. Das folgende Bild sampelt 450 Datenpixel auf 530 gerenderte Pixel hoch. Möglicherweise bemerken Sie ein Gitter von linienartigen Artefakten, die von den zusätzlichen Pixeln stammen, die erstellt werden mussten. Da die Interpolation 'nearest' ist, sind sie identisch mit einer benachbarten Pixelreihe und dehnen das Bild lokal so, dass es verzerrt aussieht.
fig, ax = plt.subplots(figsize=(6.8, 6.8))
ax.imshow(alarge, interpolation='nearest', cmap='grey')
ax.set_title("up-sampled by factor a 1.17, interpolation='nearest'")
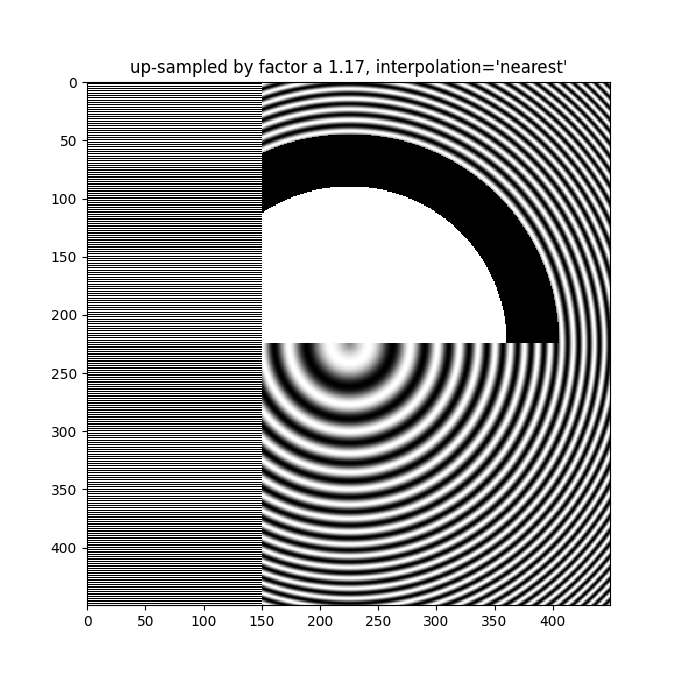
Bessere Anti-Aliasing-Algorithmen können diesen Effekt reduzieren
fig, ax = plt.subplots(figsize=(6.8, 6.8))
ax.imshow(alarge, interpolation='auto', cmap='grey')
ax.set_title("up-sampled by factor a 1.17, interpolation='auto'")
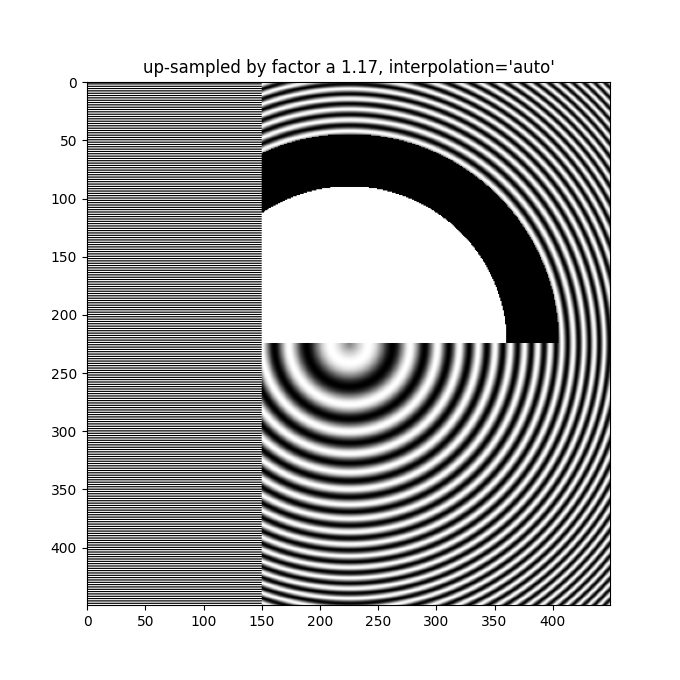
Abgesehen vom Standard 'hanning'-Anti-Aliasing unterstützt imshow eine Reihe verschiedener Interpolationsalgorithmen, die je nach zugrunde liegenden Daten besser oder schlechter funktionieren können.
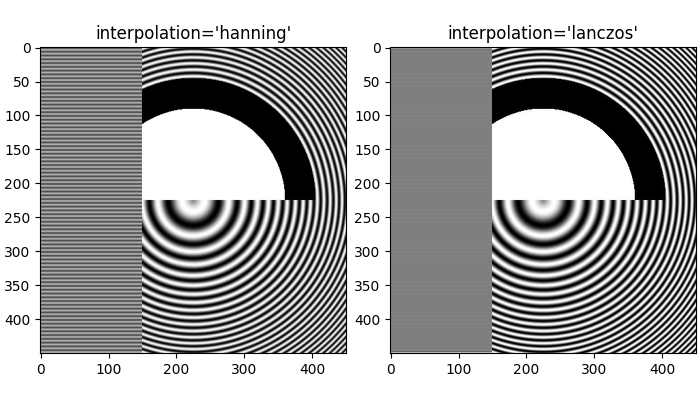
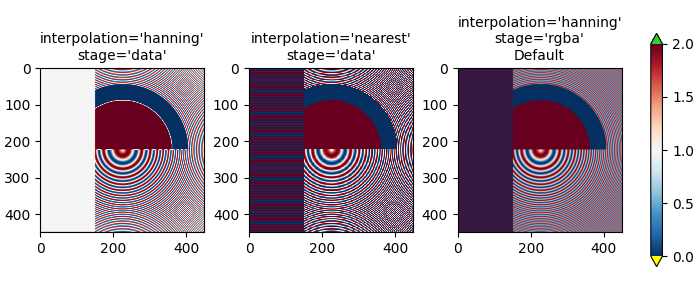
Ein letztes Beispiel zeigt die Wünschbarkeit, die Anti-Aliasing-Filterung auf der RGBA-Stufe durchzuführen, wenn nicht-triviale Interpolationskerne verwendet werden. Im Folgenden sind die Daten in den oberen 100 Zeilen exakt 0,0, und die Daten im inneren Kreis sind exakt 2,0. Wenn wir die interpolation_stage im 'data'-Raum durchführen und einen Anti-Aliasing-Filter verwenden (erstes Panel), führen Fließkommaungenauigkeiten dazu, dass einige der Datenwerte knapp unter Null oder knapp über 2,0 liegen und ihnen die Unter- oder Überfarben zugewiesen werden. Dies kann vermieden werden, wenn kein Anti-Aliasing-Filter verwendet wird (interpolation auf 'nearest' gesetzt), aber das macht den Teil der Daten, der anfällig für Moiré-Muster ist, viel schlimmer (zweites Panel). Daher empfehlen wir die Standardeinstellungen interpolation von 'hanning'/'auto' und interpolation_stage von 'rgba'/'auto' für die meisten Down-Sampling-Situationen (letztes Panel).
a = alarge + 1
cmap = plt.get_cmap('RdBu_r')
cmap.set_under('yellow')
cmap.set_over('limegreen')
fig, axs = plt.subplots(1, 3, figsize=(7, 3), layout='constrained')
for ax, interp, space in zip(axs.flat,
['hanning', 'nearest', 'hanning', ],
['data', 'data', 'rgba']):
im = ax.imshow(a, interpolation=interp, interpolation_stage=space,
cmap=cmap, vmin=0, vmax=2)
title = f"interpolation='{interp}'\nstage='{space}'"
if ax == axs[2]:
title += '\nDefault'
ax.set_title(title, fontsize='medium')
fig.colorbar(im, ax=axs, extend='both', shrink=0.8)
Up-Sampling#
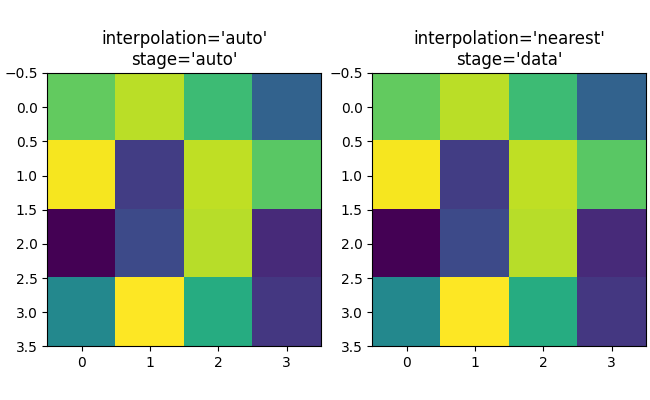
Wenn wir up-sampled, können wir ein Datenpixel durch viele Bild- oder Bildschirm-Pixel darstellen. Im folgenden Beispiel übersampeln wir die kleine Datenmatrix stark.
np.random.seed(19680801+9)
a = np.random.rand(4, 4)
fig, axs = plt.subplots(1, 2, figsize=(6.5, 4), layout='compressed')
axs[0].imshow(asmall, cmap='viridis')
axs[0].set_title("interpolation='auto'\nstage='auto'")
axs[1].imshow(asmall, cmap='viridis', interpolation="nearest",
interpolation_stage="data")
axs[1].set_title("interpolation='nearest'\nstage='data'")
plt.show()
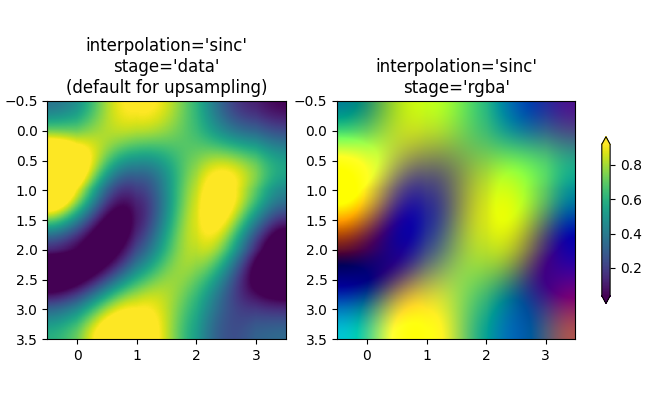
Das Schlüsselwortargument interpolation kann verwendet werden, um die Pixel bei Bedarf zu glätten. Dies ist jedoch fast immer besser im Datenraum als im RGBA-Raum durchzuführen, wo die Filter Farben erzeugen können, die nicht in der Colormap enthalten sind. Im folgenden Beispiel beachten Sie, dass bei 'rgba'-Interpolation rote Farben als Interpolationsartefakte auftreten. Daher ist die Standardwahl 'auto' für interpolation_stage gleich 'data' beim Up-Sampling von mehr als dem Faktor drei.
fig, axs = plt.subplots(1, 2, figsize=(6.5, 4), layout='compressed')
im = axs[0].imshow(a, cmap='viridis', interpolation='sinc', interpolation_stage='data')
axs[0].set_title("interpolation='sinc'\nstage='data'\n(default for upsampling)")
axs[1].imshow(a, cmap='viridis', interpolation='sinc', interpolation_stage='rgba')
axs[1].set_title("interpolation='sinc'\nstage='rgba'")
fig.colorbar(im, ax=axs, shrink=0.7, extend='both')
Vermeidung von Neuberechnung#
Es ist möglich, die Neuberechnung von Daten beim Erstellen eines Bildes zu vermeiden. Eine Methode besteht darin, einfach auf ein Vektor-Backend (pdf, eps, svg) zu speichern und interpolation='none' zu verwenden. Vektor-Backends ermöglichen eingebettete Bilder. Beachten Sie jedoch, dass einige Vektor-Bildbetrachter möglicherweise Bildpixel glätten.
Die zweite Methode besteht darin, die Größe Ihrer Achsen genau an die Größe Ihrer Daten anzupassen. Die folgende Abbildung ist genau 2 Zoll mal 2 Zoll groß, und wenn die DPI 200 beträgt, werden die 400x400 Daten überhaupt nicht neu berechnet. Wenn Sie dieses Bild herunterladen und in einem Bildbetrachter zoomen, sollten Sie die einzelnen Streifen auf der linken Seite sehen (beachten Sie, dass der HTML-Code möglicherweise eine 100x100-Version des Bildes liefert, die down-sampled wird, wenn Sie keinen HiDPI- oder "Retina"-Bildschirm haben).
fig = plt.figure(figsize=(2, 2))
ax = fig.add_axes([0, 0, 1, 1])
ax.imshow(aa[:400, :400], cmap='RdBu_r', interpolation='nearest')
plt.show()

Referenzen
Die Verwendung der folgenden Funktionen, Methoden, Klassen und Module wird in diesem Beispiel gezeigt
Gesamtlaufzeit des Skripts: (0 Minuten 10,892 Sekunden)