matplotlib.pyplot.hist2d#
- matplotlib.pyplot.hist2d(x, y, bins=10, *, range=None, density=False, weights=None, cmin=None, cmax=None, data=None, **kwargs)[Quelle]#
Erstellt ein 2D-Histogramm.
- Parameter:
- x, yarray-like, shape (n, )
Eingabewerte
- binsNone oder int oder [int, int] oder array-like oder [array, array]
Die Spezifikation der Bins
Wenn int, die Anzahl der Bins für die beiden Dimensionen (
nx = ny = bins).Wenn
[int, int], die Anzahl der Bins in jeder Dimension (nx, ny = bins).Wenn array-like, die Bin-Kanten für die beiden Dimensionen (
x_edges = y_edges = bins).Wenn
[array, array], die Bin-Kanten in jeder Dimension (x_edges, y_edges = bins).
Der Standardwert ist 10.
- rangearray-like shape(2, 2), optional
Die linkesten und rechtesten Kanten der Bins entlang jeder Dimension (wenn nicht explizit in den bins-Parametern angegeben):
[[xmin, xmax], [ymin, ymax]]. Alle Werte außerhalb dieses Bereichs werden als Ausreißer betrachtet und nicht im Histogramm gezählt.- densitybool, Standard: False
Normalisiert das Histogramm. Details finden Sie in der Dokumentation des density-Parameters von
hist.- weightsarray-like, shape (n, ), optional
Ein Array von Werten w_i, die jede Stichprobe (x_i, y_i) gewichten.
- cmin, cmaxfloat, default: None
Alle Bins, deren Anzahl kleiner als cmin oder größer als cmax ist, werden nicht angezeigt (vor der Übergabe an
pcolormeshauf NaN gesetzt) und diese Zählwerte im zurückgegebenen Zählhistogramm werden ebenfalls auf NaN gesetzt.
- Gibt zurück:
- h2D array
Das zweidimensionale Histogramm der Stichproben x und y. Werte in x werden entlang der ersten Dimension histogrammiert und Werte in y entlang der zweiten Dimension.
- xedges1D array
Die Bin-Kanten entlang der x-Achse.
- yedges1D array
Die Bin-Kanten entlang der y-Achse.
- image
QuadMesh
- Andere Parameter:
- cmapstr oder
Colormap, Standard:rcParams["image.cmap"](Standard:'viridis') Die Colormap-Instanz oder der registrierte Colormap-Name, der verwendet wird, um Skalardaten in Farben abzubilden.
- normstr oder
Normalize, optional Die Normalisierungsmethode, die verwendet wird, um Skalardaten vor der Abbildung auf Farben mithilfe von cmap in den Bereich [0, 1] zu skalieren. Standardmäßig wird eine lineare Skalierung verwendet, die den niedrigsten Wert auf 0 und den höchsten auf 1 abbildet.
Wenn angegeben, kann dies eine der folgenden Optionen sein
Eine Instanz von
Normalizeoder eine ihrer Unterklassen (siehe Farbkartennormalisierung).Ein Skalennamen, d. h. einer von "linear", "log", "symlog", "logit" usw. Für eine Liste der verfügbaren Skalen rufen Sie
matplotlib.scale.get_scale_names()auf. In diesem Fall wird eine geeigneteNormalize-Unterklasse dynamisch generiert und instanziiert.
- vmin, vmaxfloat, optional
Wenn Skalardaten und keine explizite norm verwendet werden, definieren vmin und vmax den Datenbereich, den die Farbkarte abdeckt. Standardmäßig deckt die Farbkarte den gesamten Wertebereich der bereitgestellten Daten ab. Es ist ein Fehler, vmin/vmax zu verwenden, wenn eine norm-Instanz angegeben ist (aber die Verwendung eines
str-norm-Namens zusammen mit vmin/vmax ist akzeptabel).- colorizer
Colorizeroder None, Standard: None Das Colorizer-Objekt, das verwendet wird, um Farbe auf Daten abzubilden. Wenn None, wird ein Colorizer-Objekt aus norm und cmap erstellt.
- alpha
0 <= scalar <= 1oderNone, optional Der Alpha-Mischwert.
- dataindizierbares Objekt, optional
Wenn angegeben, akzeptieren die folgenden Parameter auch einen String
s, der alsdata[s]interpretiert wird, wennsein Schlüssel indataistx, y, weights
- **kwargs
Zusätzliche Parameter werden an die Methode
pcolormeshund den KonstruktorQuadMeshweitergegeben.
- cmapstr oder
Anmerkungen
Hinweis
Dies ist der pyplot-Wrapper für
axes.Axes.hist2d.Derzeit berechnet
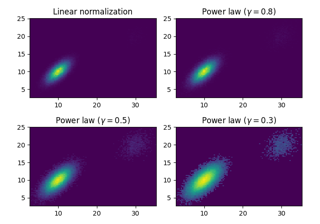
hist2dseine eigenen Achsenlimits, und alle zuvor gesetzten Limits werden ignoriert.Das Rendern des Histogramms mit einer logarithmischen Farbskala wird durch Übergabe einer Instanz von
colors.LogNorman das Schlüsselwortargument norm erreicht. Ebenso kann eine Potenzgesetz-Normalisierung (ähnlich der Gamma-Korrektur) mitcolors.PowerNormerreicht werden.