Erste Schritte mit mpl-probscale¶
Installation¶
mpl-probscale wird unter Python 3.6 entwickelt. Es wird auch unter Python 3.4, 3.5 und (derzeit) sogar 2.7 getestet.
Über conda¶
Offizielle Veröffentlichungen von mpl-probscale finden Sie auf conda-forge
conda install --channel=conda-forge mpl-probscale
Ziemlich aktuelle Builds der Entwicklungsversionen sind auf meinem Kanal verfügbar
conda install --channel=conda-forge mpl-probscale
Über PyPI¶
Offizielle Quellcode-Veröffentlichungen sind auch auf PyPI verfügbar pip install probscale
Aus dem Quellcode¶
mpl-probscale ist ein reines Python-Paket. Die Installation aus dem Quellcode sollte auf jeder Plattform ziemlich einfach sein. Laden Sie dazu von github herunter oder klonen Sie es, entpacken Sie das Archiv bei Bedarf und führen Sie dann aus
cd mpl-probscale # or wherever the setup.py got placed
pip install .
Ich empfehle pip install . gegenüber python setup.py install aus Gründen, die ich nicht vollständig verstehe.
%matplotlib inline
import warnings
warnings.simplefilter('ignore')
import numpy
from matplotlib import pyplot
from scipy import stats
import seaborn
clear_bkgd = {'axes.facecolor':'none', 'figure.facecolor':'none'}
seaborn.set(style='ticks', context='talk', color_codes=True, rc=clear_bkgd)
Hintergrund¶
Integrierte Matplotlib-Skalen¶
Für den Gelegenheitsnutzer können Matplotlib-Skalen entweder auf "linear" oder "log" (logarithmisch) gesetzt werden. Es gibt noch andere (z. B. logit, symlog), aber ich habe sie in der Praxis noch nicht oft gesehen.
Lineare Skalen sind der Standard
fig, ax = pyplot.subplots()
seaborn.despine(fig=fig)
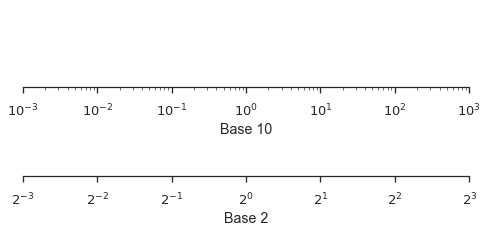
Logarithmische Skalen können gut funktionieren, wenn Ihre Daten mehrere Größenordnungen umfassen und nicht unbedingt auf Basis 10 sein müssen.
fig, (ax1, ax2) = pyplot.subplots(nrows=2, figsize=(8,3))
ax1.set_xscale('log')
ax1.set_xlim(left=1e-3, right=1e3)
ax1.set_xlabel("Base 10")
ax1.set_yticks([])
ax2.set_xscale('log', basex=2)
ax2.set_xlim(left=2**-3, right=2**3)
ax2.set_xlabel("Base 2")
ax2.set_yticks([])
seaborn.despine(fig=fig, left=True)
Wahrscheinlichkeitsskalen¶
mpl-probscale ermöglicht die Verwendung von Wahrscheinlichkeitsskalen. Alles, was Sie tun müssen, ist, es zu importieren.
Vor dem Import ist keine Wahrscheinlichkeitsskala in Matplotlib verfügbar
try:
fig, ax = pyplot.subplots()
ax.set_xscale('prob')
except ValueError as e:
pyplot.close(fig)
print(e)
Unknown scale type 'prob'
Um auf Wahrscheinlichkeitsskalen zuzugreifen, importieren Sie einfach das Modul probscale.
import probscale
fig, ax = pyplot.subplots(figsize=(8, 3))
ax.set_xscale('prob')
ax.set_xlim(left=0.5, right=99.5)
ax.set_xlabel('Normal probability scale (%)')
seaborn.despine(fig=fig)
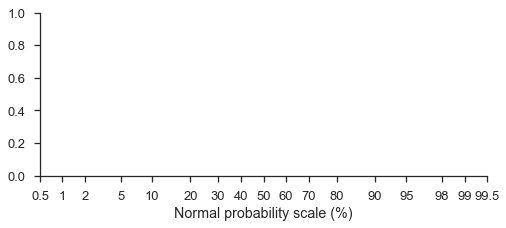
Wahrscheinlichkeitsskalen sind standardmäßig auf die Standardnormalverteilung eingestellt (beachten Sie, dass die Formatierung eine prozentbasierte Wahrscheinlichkeit ist)
Sie können sogar verschiedene Wahrscheinlichkeitsverteilungen verwenden, obwohl dies schwierig sein kann. Sie müssen eine eingefrorene Verteilung entweder aus scipy.stats oder paramnormal an das Schlüsselwortargument dist in ax.set_[x|y]scale übergeben.
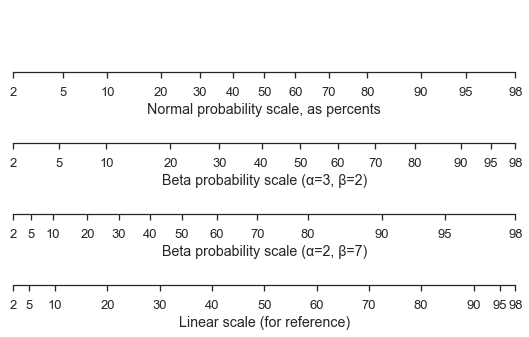
Hier ist eine Standard-Normalverteilungsskala mit zwei verschiedenen Beta-Skalen und einer linearen Skala zum Vergleich.
fig, (ax1, ax2, ax3, ax4) = pyplot.subplots(figsize=(9, 5), nrows=4)
for ax in [ax1, ax2, ax3, ax4]:
ax.set_xlim(left=2, right=98)
ax.set_yticks([])
ax1.set_xscale('prob')
ax1.set_xlabel('Normal probability scale, as percents')
beta1 = stats.beta(a=3, b=2)
ax2.set_xscale('prob', dist=beta1)
ax2.set_xlabel('Beta probability scale (α=3, β=2)')
beta2 = stats.beta(a=2, b=7)
ax3.set_xscale('prob', dist=beta2)
ax3.set_xlabel('Beta probability scale (α=2, β=7)')
ax4.set_xticks(ax1.get_xticks()[12:-12])
ax4.set_xlabel('Linear scale (for reference)')
seaborn.despine(fig=fig, left=True)
Fertige Wahrscheinlichkeitsdiagramme¶
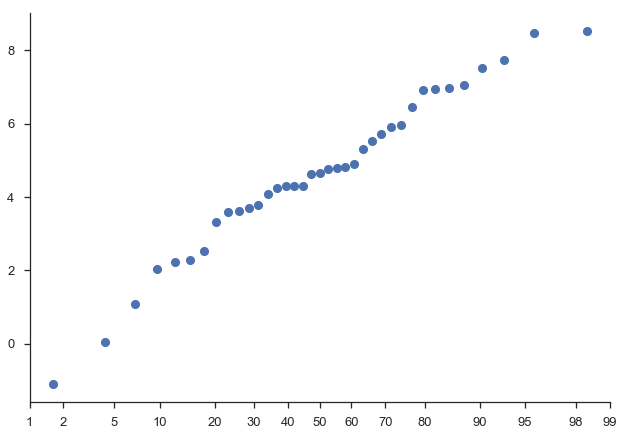
mpl-probscale wird mit einem kleinen viz Modul geliefert, das Ihnen helfen kann, ein Wahrscheinlichkeitsdiagramm einer Stichprobe zu erstellen.
Mit nur den Stichprobendaten erstellt probscale.probplot eine Abbildung, berechnet die Plotposition und die Nicht-Überschreitungswahrscheinlichkeiten und plottet alles
numpy.random.seed(0)
sample = numpy.random.normal(loc=4, scale=2, size=37)
fig = probscale.probplot(sample)
seaborn.despine(fig=fig)
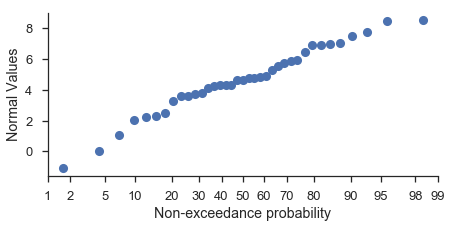
Sie sollten die Matplotlib-Achsen angeben, auf denen das Diagramm erfolgen soll, wenn Sie das Diagramm direkt mit Matplotlib-Befehlen anpassen möchten
fig, ax = pyplot.subplots(figsize=(7, 3))
probscale.probplot(sample, ax=ax)
ax.set_ylabel('Normal Values')
ax.set_xlabel('Non-exceedance probability')
ax.set_xlim(left=1, right=99)
seaborn.despine(fig=fig)
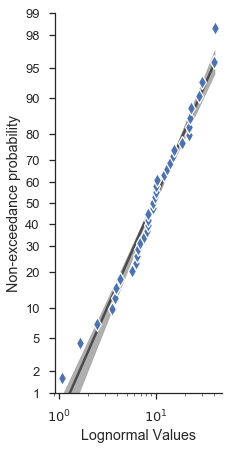
Viele weitere Optionen sind direkt aus der Signatur der Funktion probplot zugänglich.
fig, ax = pyplot.subplots(figsize=(3, 7))
numpy.random.seed(0)
new_sample = numpy.random.lognormal(mean=2.0, sigma=0.75, size=37)
probscale.probplot(
new_sample,
ax=ax,
probax='y', # flip the plot
datascale='log', # scale of the non-probability axis
bestfit=True, # draw a best-fit line
estimate_ci=True,
datalabel='Lognormal Values', # labels and markers...
problabel='Non-exceedance probability',
scatter_kws=dict(marker='d', zorder=2, mew=1.25, mec='w', markersize=10),
line_kws=dict(color='0.17', linewidth=2.5, zorder=0, alpha=0.75),
)
ax.set_ylim(bottom=1, top=99)
seaborn.despine(fig=fig)
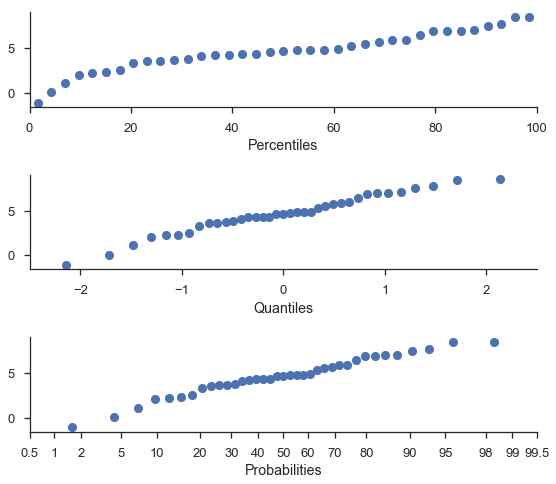
Perzentil- und Quantilsdiagramme¶
Zur Vereinfachung können Sie mit derselben Funktion Perzentil- und Quantilsdiagramme erstellen.
Hinweis
Die Perzentil- und Wahrscheinlichkeitsachsen werden gegen dieselben Werte geplottet. Der Unterschied besteht lediglich darin, dass "Perzentile" auf einer linearen Skala geplottet werden.
fig, (ax1, ax2, ax3) = pyplot.subplots(nrows=3, figsize=(8, 7))
probscale.probplot(sample, ax=ax1, plottype='pp', problabel='Percentiles')
probscale.probplot(sample, ax=ax2, plottype='qq', problabel='Quantiles')
probscale.probplot(sample, ax=ax3, plottype='prob', problabel='Probabilities')
ax2.set_xlim(left=-2.5, right=2.5)
ax3.set_xlim(left=0.5, right=99.5)
fig.tight_layout()
seaborn.despine(fig=fig)
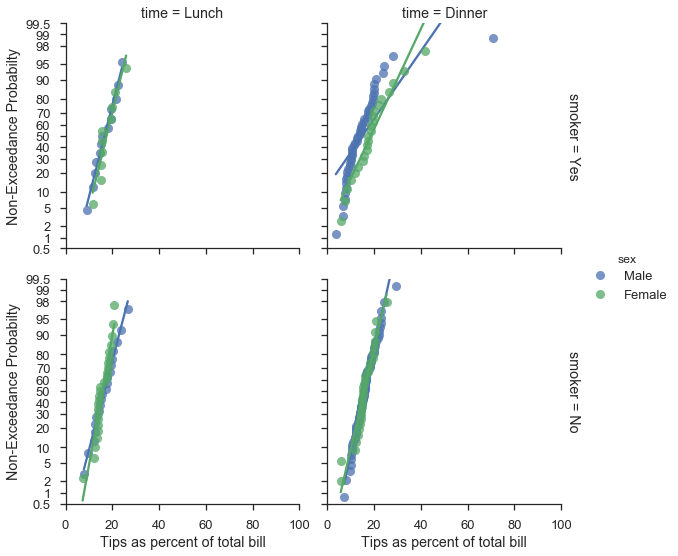
Arbeiten mit Seaborn FacetGrids¶
Gute Nachrichten für alle. Die Funktion probplot funktioniert im Allgemeinen wie erwartet mit FacetGrids.
plot = (
seaborn.load_dataset("tips")
.assign(pct=lambda df: 100 * df['tip'] / df['total_bill'])
.pipe(seaborn.FacetGrid, hue='sex', col='time', row='smoker', margin_titles=True, aspect=1., size=4)
.map(probscale.probplot, 'pct', bestfit=True, scatter_kws=dict(alpha=0.75), probax='y')
.add_legend()
.set_ylabels('Non-Exceedance Probabilty')
.set_xlabels('Tips as percent of total bill')
.set(ylim=(0.5, 99.5), xlim=(0, 100))
)